层次聚类算法之聚合聚类
层次聚类是一种无监督学习中的聚类方法,它将样本聚合到层次化的类中。层次聚类又可以分为聚合聚类(自下而上)和分裂聚类(自上而下)两种,本文主要讨论聚合聚类。
聚合聚类开始时将每个样本分到一个类,之后将相距最近的两类合并,重复此操作直到满足停止条件。
本文采用欧式距离来计算各个样本之间的距离,类间距离可以采用最短距离、最长距离和中心距离三种。
直接看代码。
导入所需库。
import numpy as np from sklearn.datasets import load_iris import matplotlib.pyplot as plt
导入数据集,数据集使用鸢尾花数据集的前一百条数据的中间两维特征。其中前50条数据属于一类,后50条属于一类,共两类。选用中间两维特征是由于这两个特征可以很清楚的区分两类数据。所以,我们的目标是把数据集分成两类。
dataSet = load_iris().data[:100,1:3] #dataSet = np.delete(dataSet,41,axis=0) size = dataSet.shape categories_number = 2
为了避免数据的某个维度的值过大而主宰了样本间距离的计算结果,将数据进行归一化处理。
#归一化 mx = np.max(dataSet,axis=0) mn = np.min(dataSet,axis=0) dataSet = (dataSet - mn) / (mx - mn)
采用欧式距离计算两两样本之间的距离,提前保存所有的计算值。
#计算所有样本点间的欧氏距离(采用最小/最大/平均距离时使用)
distance = np.zeros((size[0],size[0]))
for i in range(size[0]):
for j in range(size[0]):
distance[i,j] = np.linalg.norm(dataSet[i] - dataSet[j])
根据聚合聚类自下而上的思想,使每个样本各自成为一类。
#将每个样本点(序号)加入到一个类中
categories = []
for i in range(size[0]):
categories.append({i})
下面开始循环聚合的过程,首先判断终止条件:若剩余类别数等于所需分类的数,则停止。
while True:
#若剩余类的数量满足条件,则退出循环
if len(categories) == categories_number:
break
计算类间距离,其中可以使用三种方法计算,分别是最短距离,最长距离和中心距离。
- 最短距离:两类中最近的两个样本之间的距离。
- 最长距离:两类中最远的两个样本之间的距离。
- 中心距离:两个类别的中心之间的距离。(所有样本点各个维度的均值即为类中心)
#计算类间距离
d_c = {} #类间距离
for c1 in range(len(categories) - 1):
d_c[c1] = {}
for c2 in range(c1 + 1, len(categories)):
d_s = [] #样本间距离
def min_max_distance(flag):
#最小(flag=False)或最大(flag=True)距离
for s1 in categories[c1]:
for s2 in categories[c2]:
d_s.append(distance[s1,s2])
d_s.sort(reverse=flag)
d_c[c1][c2] = d_s[0]
def centeral_distance():
#中心距离
center_c1 = np.zeros(size[1])
for s1 in categories[c1]:
center_c1 += dataSet[s1]
center_c1 /= len(categories[c1])
center_c2 = np.zeros(size[1])
for s2 in categories[c2]:
center_c2 += dataSet[s2]
center_c2 /= len(categories[c2])
d_c[c1][c2] = np.linalg.norm(center_c1 - center_c2)
#min_max_distance(False)
centeral_distance()
合并类间距离最小的两类。
#排序找到最小的类间距离
d_c_list = []
for i in d_c.items():
for j in i[1].items():
d_c_list.append((i[0],)+j)
d_c_list.sort(key=lambda a: a[-1])
#合并类间距离最小的两个类
nearest = [d_c_list[0][0],d_c_list[0][1]]
categories[nearest[0]] = categories[nearest[0]].union(categories[nearest[1]])
categories.pop(nearest[1])
循环结束后即可输出聚类的结果。
#输出结果
for i in range(len(categories)):
print('第'+str(i+1)+'类:',categories[i])
为了更清晰的展示结果,绘制散点图。
#绘制散点图
for i in categories[0]:
plt.plot(dataSet[i][0],dataSet[i][1],'rx')
for i in categories[1]:
plt.plot(dataSet[i][0],dataSet[i][1],'bo')
plt.show()
分类结果如下:
第1类: {0,1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47,48,49}
第2类: {50,51,52,53,54,55,56,57,58,59,60,61, 62,63,64,65,66,67,68,69,70,71,72,73,74,75, 76,77,78,79,80,81,82,83,84,85,86,87,88,89, 90, 91, 92,93,94,95,96,97,98,99}
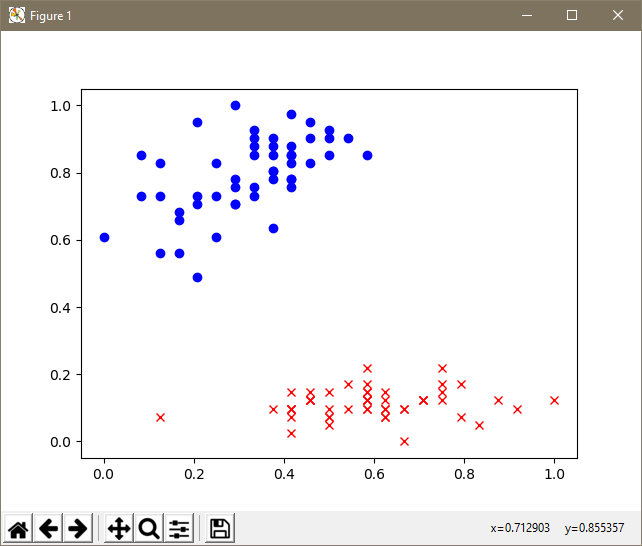
可见数据集被清楚的聚成了两类,聚类结果也是正确的。
当使用鸢尾花数据集的全部4个维度150条数据进行计算,此时结果因被分为3类。
由于4维数据难以清楚的图像化表示,因此只能取前两维绘图。
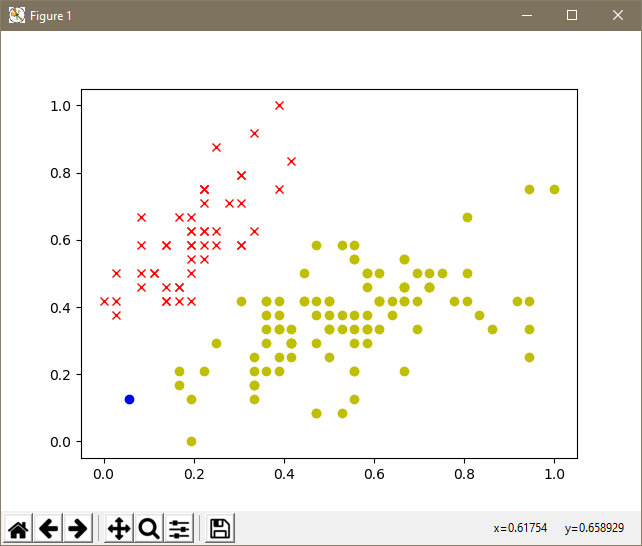
由于其中一个点可能是干扰数据或者噪音数据,其欧式距离较其他各个点更远,因此出现了某种极端的局部最优的结果,该点单独成为了一类,显然这与我们的期望是差别很大的。
聚合聚类对所有样本点间的距离都进行了细致的计算,并且聚类过程中不会进行整体的优化和二次计算,类别被不断地聚合扩充,导致很容易出现个别的噪声数据影响全局的情况发生。因此,对于距离区分不明显或含有噪音数据的数据集,聚合聚类很难很好的发挥作用,这是一大缺陷。